Genetic origin of the Swedish Sami inferred from HLA class I and
class II allele frequencies
European journal of human genetics : EJHG. 06/2008;
Samerna i norra Skandinavien är genetiska avvikare bland europeiska
populationer och deras ursprung har svårt att avgöra. För att studera den
genetiska ursprunget för de svenska samerna har vi utfört högupplösta skriva
av klass I HLA-A och-B loci och klass II DRB1, DQB1 och DQA1 loci i de norra
och södra svenska samerna. Flera av de gemensamma klass I allelerna i Sami (B
(*) 0702, B (*) 1501, B (*) 4002 och A (*) 0301) finns i hög frekvens i andra
europeiska populationer. Men ett antal av klass I och klass II alleler (B (*)
4001, A (*) 2402, DRB1 (*) 0901 och DRB1 (*) 1101) i svenska samerna är
karakteristiska för asiatiska populationer. Inblandning analyser visar att 87%
av den samiska genbank är av europeiskt ursprung och att de asiatiska bidrag
är 13%. Vår HLA-analyser visar en högre andel av asiatisk härkomst i den
samiska än vad som framgår av tidigare genetiska studies.European tidning
humangenetik förväg online offentliggörande den 14 maj 2008, doi:
10.1038/ejhg.2008.88.
European Journal of Human Genetics - A recent genetic link
between ...
Sammanfattning (Direkt översatt från engelska via Google)
Den genetiska ursprung samerna är gåtfulla och bidrag från kontinentala
Europa, Östeuropa och Asien har föreslagits. För att hantera de evolutionära
historia norra och södra Svenska samer har vi studerat deras mtDNA haplogroup
frekvenser och kompletta mtDNA genomet sekvenser. Medan de flesta av mtDNA
mångfald i norra svenska, norska och finska samerna utgörs av haplogroups V
och U5b1b1, södra svenska samerna har andra haplogroups och en frekvens
distribution liknar den kontinentala europeiska befolkningen. Stratifiering av
sydsamiska på grund av ockupationen indikerar att detta är resultatet av den
senaste tidens inblandning med den svenska befolkningen. Skillnaderna Temne
för samerna haplogroup V sekvenser är 7600 YBP (år innan nuvarande) och för
U5b1b1, 5500 YBP bland samiska och 6600 YBP bland samer och finnar. Detta
tyder på en ankomst i regionen snart efter det att glacial is, antingen genom
kontinentala Europa och / eller Volga-Ural-regionen. Haplogroup Z finns på låg
frekvens i samiska och norra asiatiska populationer, men är så gott som ute i
Europa. Flera bevarade substitutioner grupp samerna Z linjerna starkt med dem
som kommer från Finland och Volga-Ural-regionen i Ryssland, men skilja dem
från nordöstra Asien företrädare. Detta tyder på att vissa Sami linjerna
delade gemensamma förfader med linjerna från Volga-Ural området så sent som
2700 år sedan, vittnar om en nyare bidrag människor från Volga-Ural-regionen
till den samiska befolkningen.
Nyckelord: mtDNA, mitokondrier, mänskliga evolution, populationsgenetik, Sami
Till början av pageIntroduction
Samerna är ursprungsbefolkningen som lever i norra Shield, ett område som även
de nordligaste delarna av Finland, Norge, Sverige och Kolahalvön i Ryssland.
Den traditionella samiska livsstilen är nomadisk, baserat på renskötsel, fiske
och jakt. Sami är den sista europeiska befolkningen att leva ett uppehälle
livsstil och sin traditionella kost består av stora mängder av animaliska
produkter, särskilt från reindeer.1, 2 Med ökad kontakt med omgivande
befolkning livsstilen för samerna har blivit alltmer "Westernised" och många
nu bor i städer och har yrken på samma sätt som andra lokala befolkningen. Det
evolutionära ursprunget för den samiska befolkningen har ett enigma.
Arkeologiska platser i mellersta och norra Sverige visar att det finns
människor i det här området så tidigt som 9000 år före nutid (YBP). Bland de
första områden som blivit tillgängliga efter den sista NEDISNING var
Atlantkusten i Skandinavien, och människor kan ha avancerade längs norska
kusten och sedan flyttade inre mot Sverige och Finland. Människor kan också ha
kommit fram till den nordliga Shield via Finland från västra Eurasien eller
kontinentala Europa. Senaste utgrävningar i mellersta Sverige visar att
renskötsel har funnits i minst 1000 år och att den samiska befolkningen i
detta område efter Medieval.3, 4 Även människor kan ha kommit i norra Shield
snart efter sista NEDISNING, förhållandet mellan dessa tidiga bosättare och
Sami people är inte känd.
Sammanlagt 11 samiska dialekter är erkänd, men endast sex av dessa är
gemensamma användningen av de återstående fem antingen utdöda eller talat med
mindre än 50 personer. De samiska språken är medlemmar i Finnic grupp inom den
finsk-ugriska subfamily i Uralic språk. Bortsett från det samiska, det Finnic
gruppen består av språk från Finland och Estland samt språk flera
ursprungsbefolkningar från västra Ryssland, inklusive Udmurtiska, Mari och
Komi, kring korsningen av Volga och Kama vattendrag som ligger väster om Ural
bergen.
Tidiga studier visade att frekvensen av blod och protein polymorfismer
varierar avsevärt mellan samerna och den allmänna svenska population5 och för
vissa loci uppgifterna har tolkats som ett tecken på en asiatisk influence.6
På grundval av klassisk markörer, det samiska kluster med andra kaukasiska
befolkningen utan som en avvikare till kontinentaleuropeiska populations.7, 8
Undersökningar av flera DNA-markörer har bekräftat den totala likheten mellan
samiska med andra europeiska populationer, 9, men vissa genetiska markörer ger
resultat som överensstämmer med en genetisk bidrag från asiatiska
populationer, 10 särskiljande Sami från folken i Syd-och Västeuropa.
Y-kromosom haplotypes har pekat på flera grundläggande linjerna i både
finländare och samer och asiatiska inslag hos män har beräknats till cirka
50%.
Mitokondrie-DNA (mtDNA) är ofta används för befolkningen studier. Dessa
studier har utnyttjat det faktum att mtDNA är alla praktiska ändamål, kloner
ärvda och att den uppvisar en hög grad av polymorfism. Även senare databas
resurser möjliggör jämförelser av de mönster av genetisk variation i hela
mtDNA arvsmassan i stora befolkningen samples.11 Två mtDNA haplogroups
(betecknas V och U5b) 12, 13 står för majoriteten av mtDNA mångfald i norska,
finska och norra svenska Sami, med ett fåtal andra haplogroups (H, Z och D5)
uppstår vid mycket lägre frequencies.14 Haplogroup V finns i hela Europa och i
låga frekvenser i Östeuropa populations14 och har den högsta frekvensen i
svenska (68%) i förhållande till finska ( 37%) och norska samiska (33%).
Haplogroup U5b är närvarande vid låga frekvenser över Europe15 och visar en
motsatt utveckling med 26% i svenska, 41% i finska och 57% i norska Sami.14 De
allra flesta samiska U5b sekvenser bära HVR-I (hypervariable region 1)
substitutioner vid 16144, 16189 och 16270 som var kallad Sami-specifika
motif'8 eller U5b115 haplogroup. Vidare kodande regionen variation har minskat
denna beteckning till U5b1b (ntps 7385 och 10927) och med tillägg av
övergången till 16144 för "Sami-specifika" subclade har betecknas U5b1b1.14
Haplogroup H finns på låg frekvens i samernas förhållande till andra nordiska
eller kontinentala europeiska populations.14, 16 Haplogroup H är den mest
gemensamma haplogroup i EU: s befolkningar och är närvarande vid låga
frekvenser i Volga-Finnic populationer och sällsynta i centralasiatiska
befolkningen. 14, 17 Den troliga europeiskt ursprung och högre frekvens av H
bland norska samiska föreslår att haplogroup H in Fennoscandia via en
migration längs Atlantkusten i Norge eller kan förekomma i samiska på grund av
nyare inblandning av Europeiska populations.14 Återstående haplogroups
återfinns på uppskattbar frekvenser i Sami är D5 och Z.14 Medan D5 och Z
befinner sig på låga frekvenser i vissa asiatiska populationer och D5 är
relativt vanligt i Kina, 18 båda är så gott som ute i Europa, vilket innebär
ett asiatiskt ursprung. Haplogroup Z är vanligast i Nordöstra Asia19 och i
sibiriska befolkningar liksom i Volga-Ural region.14 Medan subhaplogroup Z120,
21 har observerats i Koryak och Itelmen populationer, 19 har det också noteras
att redovisa för alla Z linjerna i Västasien och norra Europe.14
Mönstret för mtDNA haplogroup frekvenser i samiska visar att befolkningen kan
ha påverkats av flera migrationer från olika källor populationer. Tidigare
studier av mitokondrie-DNA från Sami har fokuserat på haplogroup frekvenser
och uppskattningar av den genetiska mångfalden bygger på HVR-sekvenser. Här
presenterar vi en analys av kompletta mtDNA arvsmassa från norra och södra
Svenska samiska grupper, i syfte att studera den genetiska strukturen hos
populationer och behandlar ursprunget för Sami people.
Till början av pageMaterials och metoder
Befolkning prover
Frekvensen av mitokondriell haplogroups bestämdes genom följande befolkningen
prov, svenska samerna från Norrbottens län, betecknas norra svenska Sami (n =
152) och svenska samerna från Västerbottens Län, betecknas södra svenska Sami
(n = 138). Haplogroup frekvenser från andra populationer erhölls från Human
Mitochondrial Genome Database (mtDB) 11 och offentliggöras data.14 Hela mtDNA
genomet fastställdes för 18 personer från norra svenska Sami (n = 7), södra
svenska Sami (n = 7) och Volga-Ural-regionen (n = 4) (tidigare bestämt sig för
att bära Z1 subhaplogroup). Den 18 mitokondrie-sekvenser fram för denna studie
(anslutningsakt nos: DQ902694 till DQ902711) och 107 publicerade sekvenser som
används för jämförelse (anslutningsakt nos: AF346988, AF347006, AP008305,
AP008381, AP008419, AP008426, AP008553, AP008756, AP008829, AP008841,
AY195750, AY195761 , AY195781, AY255155, AY339433 - AY339459, AY339514 -
AY339522, AY339530 - AY339544, AY495109, AY495118, AY495306 - AY495315,
AY495317 - AY495330, AY519493, AY713979, AY738946, AY738947, AY882400 -
AY882406, AY882409 - AY882411, AY882413 - AY882415) är finns på GenBank.
SNP-typning och DNA-sekvensering
Det mtDNA haplogroups i södra och norra svenska Sami studerades genom sekvens
analys av utvalda webbplatser som definierar de stora samiska haplogroups
U5b1b1, V, Z, D5 och H. De områden studerats omfattar NPS 4580, 7028, 7385,
10397, 12618, 12930 , 16144, 16189, 16224, 16260, 16270, 16298, 16362.
Ytterligare områden undersökts när det behövs för att lösa andra haplogroups.
Detta och Genomsekvensen sekvenseringen utfördes med hjälp primrar beskrivs av
Rieder et al22 och BigDye kemi (Applied Biosystems, CA, USA). Sekvensering
utfördes på en ABI3700 automatiserade fragment analys maskin och analyseras
med DNA-sekvensering Analys programvara (Applied Biosystems, CA, USA).
Sequence anpassning genererades med hjälp Sequencher (Gene Codes Corporation,
Ann Arbor, MI, USA) programvara.
Genetiska analyser
Alla nukleotid positioner ges är i förhållande till CRS.23 Nukleotid
skillnader mellan de olika linjerna i olika regioner beräknades med hjälp av
DnaSP software.24 ålder haplogroups uppskattades genom att bygga
Neighbour-Joining25 träd med PAUP, 26 med hjälp av Kimura 2 parameter modell
av nucleotide substitution , 27 och beräkna medelvärdet filial längder till
delade noder. I dessa beräkningar vi utgå från en konstant ersättning på 1,71
10-8 substitutioner per plats per year.28 Median gå nätverk har beräknats med
hjälp av programvaran Network 4.1.1.2 (http://www.fluxus-engineering.com). Den
inblandning analyser utfördes med hjälp av LEA (Sannolikhet bygger
uppskattningen av inblandning) programvara, med den metod som beskrivs av
Chikhi et al29 som finns på webbplatsen: http://www.cnrs-gif.fr/pge/bioinfo/lea
.
Till början av pageResults
mtDNA haplogroup frekvenser
Haplogroups V (58. 6%) och U5b (35,5%) dominerar i norra svenska samerna, med
flera andra haplogroups (H, Z) som förekommer i låg frekvens (tabell 1). Denna
fördelning är mycket likt det som presenterats tidigare för en befolkning prov
från detta område, samt till distribution i finska och norska Sami (Tabell 1)
.14 Haplogroups V och U5b förekommer också i hög frekvens i de södra svenska
samiska, tillsammans med haplogroups H, Z och en rad andra haplogroups (tabell
1). Även förekomsten av V och U5b i både norra och södra Svenska Sami på
uppskattbar frekvenser tyder på att dessa två populationer av samma genetiska
ursprung, haplogroup distribution i södra svenska samiska befolkningen skiljer
sig från den nordsamiska i flera avseenden. Frekvensen av haplogroups V och
U5b är lägre och haplogroup H (34,8%) betydligt högre i de södra svenska
samerna. Även de södra svenska samerna har ett antal andra haplogroups som
inte finns i övriga samiska befolkningen (I, J, K), men kännetecknande för
kontinentala europeiska populationer (tabell 1). Skillnaden i haplogroup
frekvens mellan södra svenska samerna och de andra samiska befolkningen kan
bero på de senaste inblandning av svenska eller andra Continental
populationer. För att ytterligare studera dessa alternativ, vi stratifierat
södra svenska Sami provet i de traditionella yrken (dvs. renskötsel) och de
med icke traditionella yrken, på antagandet att de med traditionella yrken är
mer benägna att ha enbart samiska förfäder. Den renskötare har en haplogroup
distribution liknande den i norra svenska samerna, med en lägre frekvens av
haplogroup H och en högre frekvens av V och U5b1b1 (tabell 1). Den sydsamiska
med icke traditionella yrken har en haplogroup distribution liknar den
kontinentala europeiska befolkningen, med en hög frekvens av H och andra
"icke-samiska" haplogroups utgör mer än 70%. Skillnaden mellan dessa två
grupper av södra svenska samerna kan bero på inblandning. Använda haplogroup
frekvenser för nordsamiska och Continental européer som två källa befolkning,
vi uppskattade omfattningen av inblandning från den kontinentala europeiska
befolkningen i Kombinerade södra svenska samiska vara 48%, bland dem med
traditionella yrken vara 16% och bland personer med icke traditionella yrken
som skall 67%, med hjälp av LEA programvara.
Tabell 1 - Haplogroup frekvenser i olika populations.Full tabellen
Fylogeni europeiska och östasiatiska mtDNA linjerna
Att studera sambandet och den genetiska mångfalden inom några av mtDNA
haplogroups i samiska befolkningen, vi sekvenserade komplett Sami
mitochondrial genomer från varje haplogroups V, U5b1b1 och Z och kompletteras
våra datauppsättning med publicerade Sami sekvenser och kompletta sekvenser
från andra populationer. Median-Joining nätverk byggda för var och en av dessa
haplogroups att studera sambandet mellan den samiska mtDNA sekvenser och
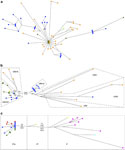
sekvenser från andra populationer. I nätverket för haplogroup V, Sami mtDNA
sekvenser är utspridda och främst gruppen med sekvenser från Finland (figur
1a). Tre av de samiska har identiska sekvenser men det finns ingen indikation
på monophyletic grupper av Sami sekvenser. Nätverket för haplogroup U5b
innehåller företrädare för subhaplogroups U5b1b, U5b1b1, U5b1a, U5b1, U5b2 och
U5b (figur 1b). Alla samiska sekvenser finns i U5b1b1 clade tillsammans med
några sekvenser från Finland. Den nära relation med sekvenser från Finland kan
bero på admixture.30 Den nukleotid mångfald Sami sekvenser av haplogroups
U5b1b1 och V är mycket lågt (= 1 10-4 och = 1.8 10-4 respektive). Beräknas
utifrån medelvärdet filial längd till deras gemensamma noden, den tid de
senaste gemensamma förfadern för samisk haplogroup V sekvenser är 7600 YBP och
för U5b1b1 5500 YBP bland samiska och 6600 YBP bland samer och finnar. Den
uppskattade åldern på U5b1b1 clades i allmänhet med en uppskattning av åldern
på sina fäderneärvda haplogroup (U5b1b) av 8600 YBP.31
Figur 1.Median gå nätverk med relevant ersättning positioner baseras på den
kompletta mtDNA genomet sekvenser för enskilda personer med haplogroups (a) V
(b) U5b (c) Z. Nätverken har byggts som beskrivs i texten. Den färgkodning är
följande: grön - samiska, blå - Finland, röd - Volga-Ural, orange -
Continental Europa, rosa - Japan, gul - NE Asia, ljusblå - Kina, ljusgrönt -
Indien.
All siffra och legend (162K)
Med undantag för en Jakutiska sekvens som hör till haplogroup U5b den enda
asiatiska sekvenser som delar en nära relation med Sami sekvenser är medlemmar
i haplogroup Z, som omfattar östra Asien och Eurasien linjerna. Nätverket för
haplogroup Z visar en tydlig Z1 sekvenser mellan finnar, samer, Volga-Ural och
en Koryak från östasiatiska Z sekvenser (figur 1c). De genetiska skillnaderna
mellan Koryak och Sami, finländare och Volga-Ural-sekvenser (fast kodande
regionen substitutioner vid ntps 740, 9494, 12930) motiverar utnämningen av
dessa sekvenser som en särskild undergrupp, betecknas Z1a. Kodningen region
nukleotid mångfalden inom Z1a grupp är anmärkningsvärt lågt (= 7,1 10-5),
vilket tyder på att dessa 18 sekvenser från tre populationer sista delade
gemensamma förfader helt nyligen. Beräknas utifrån medelvärdet filial längden
på deras gemensamma noden, den senaste gemensamma förfadern till Z1a grupp
uppskattas till 2700 YBP. Däremot har den genetiska länken Z1a till Z1 i
nordöstra Asien (Koryak) sträcker sig tillbaka till 13 000 YBP.
Till början av pageDiscussion
Norra svenska samerna har två dominerande mtDNA haplogroups, liknande andra
samiska befolkningen. Förekomsten av dessa två haplogroups i alla samiska
befolkningen, om än i olika frekvenser, pekar på ett gemensamt ursprung för
alla samiska befolkningen i norra Shield område. Bland de samiska, södra
svenska samerna är avvikare i sin fördelning av mtDNA haplogroups. Den höga
frekvensen av haplogroups i kontinentala Europa i södra svenska samerna med
icke-traditionella yrken indirekt stöder inblandning med den (europeiska)
svenska befolkningen. Den inblandning analysen bekräftar denna iakttagelse ge
något stöd för den södra svenska samerna har ett annat genetiskt ursprung än
den nordsamiska.
Den samtida svenska samiska befolkningen uppskattas till ca 50 000 personer,
32 men populationsstorleken sannolikt ha varit betydligt mindre i historiska
tider. Den nära fullständig dominans av endast två haplogroups i norra
svenska, finska och norska samiska och små befolkningsstorlek visar att
samerna skulle ha varit utsatt för stark genetisk drift. Denna begränsade
populationsstorleken stöds av hög Linkage disequilibrium (LD) mellan
microsatellite och SNP-markörer i Svenska samernas förhållande till den
allmänna befolkningen i Finland och Sweden.33, 34, 35, 36
Fördelningen av Sami linjer inom Europeiska haplogroup V visar att samerna har
drabbats av en migration av Continental europeiska stammar antingen gå direkt
norrut genom Sverige eller genom Atlantkusten, alternativt via
Volga-Ural-regionen i Ryssland där V har konstaterats vid märkbar
frequencies.14 Haplogroup U5b är vida spridd i Europa och därför ger några
ledtrådar till förmodade invandring. Men U5b1b1 har en begränsad geografisk
spridning, inriktat på Nord-och Östeuropa, där det har också konstaterats i
Volga-Ural region.14 Förekomsten av haplogroup Z innebär ett bidrag, om än
begränsad, att den samiska genpool från Asien. Det nära sambandet mellan Z1a
linjerna från finländarna och samiska med dem i Volga-Ural igen förenad med
att regionen som en trolig källa för Sami mitochondrial mångfald. Det finns
dock en skillnad i den synbara åldrar för de olika samiska haplogroups. Den
nukleotid mångfald bland samiska sekvenser för de tre haplogroups studerat här
är mycket låg. Den åldrar av variationen för U5b1b1 och V bland svenska Sami
liknar (5500 och 7600 YBP respektive) men betydligt äldre än Z (2700 YBP). Den
överraskande nära koppling mellan haplogroup Z1a bland samiska och
Volga-Ural-sekvenser visar att detta haplogroup kom in under de senaste 2-3000
YBP. Våra data stöder att en migration från Östeuropa, i närheten av
Volga-Ural-regionen, är den troliga källan till mycket av den samiska mtDNA
diversity14 men anger flera migration, den första är 6-7000 YBP och minst en
ytterligare migration 2 -3000 YBP. Med tanke på likheten mellan samiska och
finska mitochondrial linjer, denna observation av flera migration händelser
skulle även stödja tidigare befolkningen genetiska studier som har visat
dubbla ursprung i den finska people.37
Abstract
The genetic origin of the Sami is enigmatic and
contributions from Continental Europe, Eastern Europe and Asia have been
proposed. To address the evolutionary history of northern and southern
Swedish Sami, we have studied their mtDNA haplogroup frequencies and
complete mtDNA genome sequences. While the majority of mtDNA diversity in
the northern Swedish, Norwegian and Finnish Sami is accounted for by
haplogroups V and U5b1b1, the southern Swedish Sami have other haplogroups
and a frequency distribution similar to that of the Continental European
population. Stratification of the southern Sami on the basis of occupation
indicates that this is the result of recent admixture with the Swedish
population. The divergence time for the Sami haplogroup V sequences is 7600
YBP (years before present), and for U5b1b1, 5500 YBP amongst Sami and 6600
YBP amongst Sami and Finns. This suggests an arrival in the region soon
after the retreat of the glacial ice, either by way of Continental Europe
and/or the Volga-Ural region. Haplogroup Z is found at low frequency in the
Sami and Northern Asian populations but is virtually absent in Europe.
Several conserved substitutions group the Sami Z lineages strongly with
those from Finland and the Volga-Ural region of Russia, but distinguish them
from Northeast Asian representatives. This suggests that some Sami lineages
shared a common ancestor with lineages from the Volga-Ural region as
recently as 2700 years ago, indicative of a more recent contribution of
people from the Volga-Ural region to the Sami population.
Keywords:
mtDNA, mitochondria, human evolution, population
genetics, Sami
Top of page
Introduction
The Sami are indigenous people inhabiting the Northern
Shield, an area including the northernmost parts of Finland, Norway,
Sweden and the Kola Peninsula of Russia. The traditional Sami lifestyle is
nomadic, based on reindeer herding, fishing and hunting. Sami are the last
European population leading a subsistence lifestyle and their traditional
diet consists of high amounts of animal products, particularly from
reindeer.1, 2 With
increased contact with surrounding populations, the lifestyle of the Sami
has become increasingly 'Westernised' and many now live in towns and have
occupations similar to other local populations. The evolutionary origin of
the Sami population has been an enigma. Archaeological sites in central
and northern Sweden reveal the presence of people in this area as early as
9000 years before present (YBP). Among the first areas to become
accessible after the last glaciation was the Atlantic coast of
Scandinavia, and humans may have advanced along the Norwegian coastline
and then moved inland towards Sweden and Finland. Humans may have also
arrived at the Northern Shield through Finland from Western Eurasia or
Continental Europe. Recent excavations in middle Sweden indicate that
reindeer herding has existed for at least 1000 years and that the Sami
population in this area is post-Medieval.3,
4 While humans may have arrived in the Northern
Shield soon after the last glaciation, the relationship between those
early settlers and the Sami people is not known.
A total of 11 Sami dialects are recognised but only six of
these are in common use with the remaining five being either extinct or
spoken by less than 50 people. The Sami languages are members of the
Finnic group, within the Finno-Ugric subfamily of the Uralic languages.
Apart from the Sami, the Finnic group comprises languages from Finland and
Estonia as well as the languages of several indigenous peoples from
western Russia, including Udmurt, Mari and Komi, centred around the
junction of the Volga and Kama rivers to the west of the Ural mountains.
Early studies showed that the frequency of blood group and
protein polymorphisms differ significantly between Sami and the general
Swedish population5 and for some loci the
data have been interpreted as indicative of an Asian influence.6
On the basis of classical markers, the Sami cluster with other Caucasian
populations but as an outlier to Continental European populations.7,
8 Studies of multiple DNA markers have confirmed
the overall similarity of Sami with other European populations,9
but some genetic markers yield results that are consistent with a genetic
contribution from Asian populations,10
distinguishing Sami from the peoples of Southern and Western Europe. Y
chromosome haplotypes have pointed to multiple founding lineages in both
Finns and Sami and the Asian component in males has been estimated to be
around 50%.
Mitochondrial DNA (mtDNA) is frequently used for
population studies. These studies have benefited from the fact that mtDNA
is, for all practical purposes, clonally inherited and that it exhibits a
high degree of polymorphism. Also, recent database resources enable
comparisons of the pattern of genetic variation in the entire mtDNA genome
in large population samples.11 Two mtDNA
haplogroups (denoted V and U5b)12,
13 account for the majority of mtDNA diversity
in Norwegian, Finnish and northern Swedish Sami, with a few other
haplogroups (H, Z and D5) occurring at much lower frequencies.14
Haplogroup V is found across Europe and in low frequencies in Eastern
European populations14 and has the highest
frequency in Swedish (68%) relative to Finnish (37%) and Norwegian Sami
(33%). Haplogroup U5b is present at low frequencies across Europe15
and shows the opposite trend with 26% in Swedish, 41% in Finnish and 57%
in Norwegian Sami.14 The vast majority of
Sami U5b sequences carry HVR-I (hypervariable region 1) substitutions at
16144, 16189 and 16270 which was referred to as the 'Sami-specific motif'8
or U5b115 haplogroup. Further, coding
region variation has narrowed this designation down to U5b1b (ntps 7385
and 10927) and with the addition of the transition at 16144 the 'Sami-specific'
subclade has been denoted U5b1b1.14
Haplogroup H is found at low frequency in the Sami
relative to other Northern or Continental European populations.14,
16 Haplogroup H is the most common haplogroup
in European populations and is present at low frequencies in Volga-Finnic
populations and rare in central Asian populations.14,
17 The probable European origin and the higher
frequency of H among Norwegian Sami suggests that haplogroup H entered
Fennoscandia via a migration along the Atlantic coast of Norway or may be
present in the Sami due to more recent admixture with European
populations.14 The remaining haplogroups
found at appreciable frequencies in Sami are D5 and Z.14
While D5 and Z are present at low frequencies in some Asian populations
and D5 is relatively common in China,18
both are virtually absent in Europe, implying an Asian origin. Haplogroup
Z is most frequent in Northeastern Asia19
and present in Siberian populations as well as in the Volga-Ural region.14
While subhaplogroup Z120, 21
has been observed in the Koryak and Itelmen populations,19
it has also been noted to account for all Z lineages in Western Asia and
Northern Europe.14
The pattern of mtDNA haplogroup frequencies in the Sami
indicates that the population may have been influenced by several
migrations from different source populations. Previous studies of the
mitochondrial DNA of the Sami have focused on haplogroup frequencies and
estimates of genetic diversity based on HVR sequences. Here, we present an
analysis of the complete mtDNA genome from the northern and southern
Swedish Sami groups, with the purpose of studying the genetic structure of
the populations and addressing the origin of the Sami people.
Top of page
Materials and methods
Population samples
The frequency of mitochondrial haplogroups were
determined from the following population samples; Swedish Sami from the
County of Norrbotten, denoted northern Swedish Sami (n=152) and
Swedish Sami from Västerbotten, denoted southern Swedish Sami (n=138).
Haplogroup frequencies from other populations were obtained from the Human
Mitochondrial Genome Database (mtDB)11 and
published data.14 The complete mtDNA
genome was determined for 18 individuals from northern Swedish Sami (n=7),
southern Swedish Sami (n=7) and the Volga-Ural region (n=4)
(previously determined to carry the Z1 subhaplogroup). The 18
mitochondrial sequences produced for this study (Accession nos:
DQ902694 to DQ902711) and 107 published sequences used for comparison
(Accession nos:
AF346988, AF347006, AP008305, AP008381, AP008419, AP008426, AP008553,
AP008756, AP008829, AP008841, AY195750, AY195761, AY195781, AY255155,
AY339433 – AY339459, AY339514 – AY339522, AY339530 – AY339544, AY495109,
AY495118, AY495306 – AY495315, AY495317 – AY495330, AY519493, AY713979,
AY738946, AY738947, AY882400 – AY882406, AY882409 – AY882411, AY882413 –
AY882415) are available at GenBank.
SNP typing and DNA sequencing
The mtDNA haplogroups of the southern and northern
Swedish Sami were studied by sequence analysis of selected sites that
define the major Sami haplogroups U5b1b1, V, Z, D5 and H. The sites
studied include nps 4580, 7028, 7385, 10397, 12618, 12930, 16144, 16189,
16224, 16260, 16270, 16298, 16362. Additional sites were examined when
necessary to resolve other haplogroups. This and complete genome
sequencing were performed using primers described by Rieder et al22
and BigDye chemistry (Applied Biosystems, CA, USA). Sequencing was
performed on an ABI3700 automated fragment analysis machine and analysed
with DNA Sequencing Analysis software (Applied Biosystems, CA, USA).
Sequence alignments were generated using Sequencher (Gene Codes
Corporation, Ann Arbor, MI, USA) software.
Genetic analyses
All nucleotide positions given are relative to CRS.23
Nucleotide diversity among lineages of various regions was estimated using
the DnaSP software.24 The age of
haplogroups was estimated by building Neighbour-Joining25
trees with PAUP,26 using the Kimura 2
parameter model of nucleotide substitution,27
and calculating the mean branch lengths to shared nodes. In these
calculations we assumed a constant substitution rate of 1.71
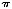 10-8 substitutions per site per year.28
Median-Joining networks were calculated using the software Network 4.1.1.2
(http://www.fluxus-engineering.com).
The admixture analyses were carried out using the LEA (Likelihood based
estimation of admixture) software, using the method described by Chikhi
et al29 that is available at the site:
http://www.cnrs-gif.fr/pge/bioinfo/lea.
10-8 substitutions per site per year.28
Median-Joining networks were calculated using the software Network 4.1.1.2
(http://www.fluxus-engineering.com).
The admixture analyses were carried out using the LEA (Likelihood based
estimation of admixture) software, using the method described by Chikhi
et al29 that is available at the site:
http://www.cnrs-gif.fr/pge/bioinfo/lea.
Top of page
Results
mtDNA haplogroup frequencies
Haplogroups V (58. 6%) and U5b (35.5%) predominate
in the northern Swedish Sami, with several other haplogroups (H, Z)
occurring at low frequency (Table 1). This
distribution is very similar to that presented previously for a population
sample from this area, as well as to the distribution in Finnish and
Norwegian Sami (Table 1).14
Haplogroups V and U5b are also present at high frequency in the southern
Swedish Sami, along with haplogroups H, Z and a range of other haplogroups
(Table 1). While the presence of V and U5b in both the
northern and southern Swedish Sami at appreciable frequencies indicates
that these two populations share the same genetic origin, the haplogroup
distribution in the southern Swedish Sami population differs from the
northern Sami in several respects. The frequency of haplogroups V and U5b
is lower and haplogroup H (34.8%) much higher in the southern Swedish
Sami. Also, the southern Swedish Sami have a number of other haplogroups
not found in other Sami populations (I, J, K) but characteristic of
Continental European populations (Table 1). The
difference in haplogroup frequency distribution between southern Swedish
Sami and the other Sami populations could be due to recent admixture with
Swedish or other Continental populations. To further study these
alternatives, we stratified the southern Swedish Sami sample into those
with traditional occupations (ie reindeer herding) and those with
nontraditional occupations, on the premise that those with traditional
occupations are more likely to have exclusively Sami ancestors. The
reindeer herders have a haplogroup distribution similar to that of
northern Swedish Sami, with a lower frequency of haplogroup H and a higher
frequency of V and U5b1b1 (Table 1). The southern Sami
with nontraditional occupations have a haplogroup distribution similar to
that of the Continental European population, with a high frequency of H
and other 'non-Sami' haplogroups constituting more than 70%. The
difference between these two groups of southern Swedish Sami could be due
to admixture. Using the haplogroup frequencies for the northern Sami and
Continental Europeans as the two source populations, we estimated the
extent of admixture from the Continental European population in the
combined southern Swedish Sami to be 48%, among those with traditional
occupations to be 16% and among those with nontraditional occupations to
be 67%, using the LEA software.
Phylogeny of European and East Asian mtDNA lineages
To study the relationship and genetic diversity
within some of the mtDNA haplogroups in Sami populations, we sequenced
complete Sami mitochondrial genomes from each of haplogroups V, U5b1b1 and
Z and supplemented our dataset with published Sami sequences and complete
sequences from other populations. Median-Joining networks were constructed
for each of these haplogroups to study the relationship of the Sami mtDNA
sequences and sequences from other populations. In the network for
haplogroup V, Sami mtDNA sequences are scattered and mainly group with
sequences from Finland (Figure 1a). Three of the Sami
have identical sequences but there is no indication of monophyletic groups
of Sami sequences. The network for haplogroup U5b contains representatives
of the subhaplogroups U5b1b, U5b1b1, U5b1a, U5b1, U5b2 and U5b (Figure
1b). All Sami sequences are found in the U5b1b1 clade together with
some sequences from Finland. The close relationship with sequences from
Finland may be due to admixture.30 The
nucleotide diversity for Sami sequences of haplogroups U5b1b1 and V is
very low (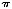 =1
=1
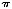 10-4 and
10-4 and
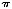 =1.8
=1.8
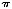 10-4, respectively). Calculated from the mean branch length to
their shared node, the time to the most recent common ancestor of Sami
haplogroup V sequences is 7600 YBP, and for U5b1b1 5500 YBP amongst Sami
and 6600 YBP among Sami and Finns. The estimated ages of the U5b1b1 clades
are in general agreement with an estimate of the age of their ancestral
haplogroup (U5b1b) of 8600 YBP.31
10-4, respectively). Calculated from the mean branch length to
their shared node, the time to the most recent common ancestor of Sami
haplogroup V sequences is 7600 YBP, and for U5b1b1 5500 YBP amongst Sami
and 6600 YBP among Sami and Finns. The estimated ages of the U5b1b1 clades
are in general agreement with an estimate of the age of their ancestral
haplogroup (U5b1b) of 8600 YBP.31
Except for one Yakut sequence belonging to haplogroup U5b,
the only Asian sequences that share a close relationship with Sami
sequences are members of haplogroup Z, which comprises East Asian and
Eurasian lineages. The network for haplogroup Z shows a clear separation
of Z1 sequences between Finns, Sami, Volga-Ural and one Koryak from East
Asian Z sequences (Figure 1c). The genetic divergence
between the Koryak and the Sami, Finns and Volga-Ural sequences (fixed
coding region substitutions at ntps 740, 9494, 12930) warrants the
designation of these sequences as a separate subgroup, denoted Z1a. The
coding region nucleotide diversity within the Z1a group is remarkably low
(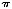 =7.1
=7.1
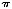 10-5), indicating that these 18 sequences from three
populations last shared a common ancestor very recently. Calculated from
the mean branch length to their common node, the most recent common
ancestor for Z1a group is estimated at 2700 YBP. By contrast, the genetic
link from Z1a to Z1 in Northeast Asia (Koryak) extends back to 13 000 YBP.
10-5), indicating that these 18 sequences from three
populations last shared a common ancestor very recently. Calculated from
the mean branch length to their common node, the most recent common
ancestor for Z1a group is estimated at 2700 YBP. By contrast, the genetic
link from Z1a to Z1 in Northeast Asia (Koryak) extends back to 13 000 YBP.
Top of page
Discussion
The northern Swedish Sami have two dominating mtDNA
haplogroups, similar to other Sami populations. The presence of these two
haplogroups in all Sami populations, albeit at different frequencies,
points to a common origin for all Sami populations in the northern Shield
area. Among the Sami, the southern Swedish Sami are outliers in their
distribution of mtDNA haplogroups. The high frequency of the haplogroups
present in Continental Europe in the southern Swedish Sami with
non-traditional occupations indirectly supports admixture with the
(European) Swedish population. The admixture analysis confirms this
observation, lending no support for the southern Swedish Sami having a
different genetic origin than the northern Sami.
The contemporary Swedish Sami population is estimated to
number about 50 000 people,32 but the
population size is likely to have been considerably smaller in historic
times. The near complete dominance of only two haplogroups in the northern
Swedish, Finnish and Norwegian Sami and the small population size
indicates that the Sami could have been subject to strong genetic drift.
This limited population size is supported by high linkage disequilibrium
(LD) between microsatellite and SNP markers in Swedish Sami relative to
the general population in Finland and Sweden.33,
34, 35, 36
The distribution of Sami lineages within the European
haplogroup V indicates that Sami have been affected by a migration of
Continental European tribes either moving directly north through Sweden or
by way of the Atlantic coast, or alternatively, via the Volga-Ural region
of Russia where V has been found at appreciable frequencies.14
Haplogroup U5b is widely dispersed in Europe and therefore provides few
clues as to putative migrations. However, U5b1b1 has a restricted
geographic distribution centred on Northern and Eastern Europe, where it
has also been identified in the Volga-Ural region.14
The presence of haplogroup Z implies a contribution, albeit limited, to
the Sami gene pool from Asia. The close relationship of Z1a lineages from
Finns and Sami with those of the Volga-Ural again implicates that region
as a probable source for Sami mitochondrial diversity. There is, however,
a difference in the apparent ages of the different Sami haplogroups. The
nucleotide diversity among Sami sequences for the three haplogroups
studied here is very low. The ages of the variation for U5b1b1 and V among
Swedish Sami are similar (5500 and 7600 YBP, respectively) but
considerably older than for Z (2700 YBP). The surprisingly close link
between haplogroup Z1a among Sami and the Volga-Ural sequences suggest
that this haplogroup was brought in during the last 2–3000 YBP. Our data
supports that a migration from Eastern Europe, in the vicinity of the
Volga-Ural region, is the likely source for much of the Sami mtDNA
diversity14 but indicates multiple
migrations, the first being 6–7000 YBP and at least one additional
migration 2–3000 YBP. Considering the similarity observed between Sami and
Finnish mitochondrial lineages, this observation of multiple migration
events would also support previous population genetic studies that have
indicated dual origins of the Finnish people.37
![]() 10-8 substitutions per site per year.28
Median-Joining networks were calculated using the software Network 4.1.1.2
(http://www.fluxus-engineering.com).
The admixture analyses were carried out using the LEA (Likelihood based
estimation of admixture) software, using the method described by Chikhi
et al29 that is available at the site:
http://www.cnrs-gif.fr/pge/bioinfo/lea.
10-8 substitutions per site per year.28
Median-Joining networks were calculated using the software Network 4.1.1.2
(http://www.fluxus-engineering.com).
The admixture analyses were carried out using the LEA (Likelihood based
estimation of admixture) software, using the method described by Chikhi
et al29 that is available at the site:
http://www.cnrs-gif.fr/pge/bioinfo/lea.![]() =1
=1
![]() 10-4 and
10-4 and
![]() =1.8
=1.8
![]() 10-4, respectively). Calculated from the mean branch length to
their shared node, the time to the most recent common ancestor of Sami
haplogroup V sequences is 7600 YBP, and for U5b1b1 5500 YBP amongst Sami
and 6600 YBP among Sami and Finns. The estimated ages of the U5b1b1 clades
are in general agreement with an estimate of the age of their ancestral
haplogroup (U5b1b) of 8600 YBP.31
10-4, respectively). Calculated from the mean branch length to
their shared node, the time to the most recent common ancestor of Sami
haplogroup V sequences is 7600 YBP, and for U5b1b1 5500 YBP amongst Sami
and 6600 YBP among Sami and Finns. The estimated ages of the U5b1b1 clades
are in general agreement with an estimate of the age of their ancestral
haplogroup (U5b1b) of 8600 YBP.31![]() =7.1
=7.1
![]() 10-5), indicating that these 18 sequences from three
populations last shared a common ancestor very recently. Calculated from
the mean branch length to their common node, the most recent common
ancestor for Z1a group is estimated at 2700 YBP. By contrast, the genetic
link from Z1a to Z1 in Northeast Asia (Koryak) extends back to 13 000 YBP.
10-5), indicating that these 18 sequences from three
populations last shared a common ancestor very recently. Calculated from
the mean branch length to their common node, the most recent common
ancestor for Z1a group is estimated at 2700 YBP. By contrast, the genetic
link from Z1a to Z1 in Northeast Asia (Koryak) extends back to 13 000 YBP.